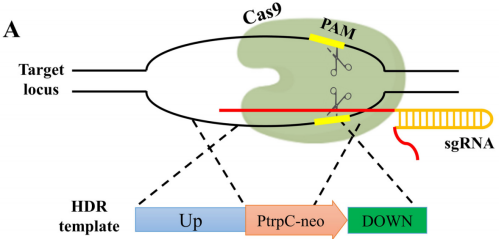
使用NEB Gibson组装试剂盒将UGI,Ptef1启动子(8)和TtrpC终止子扩增并组装到p0380-bar质粒(69)中,以形成evo-BE4max,GAM-BE4max和evo-CDA1编辑器。先前构建了Cas9表达PCR盒bar-Ptef1-Cas9-TtprC,以及选择标记neo和bar。为了选择靶向AMD,cre-1和Mtclr-2的特定sgRNA,使用sgRNACas9工具鉴定嗜热支原体基因组中的所有sgRNA靶位点,并选择高分的sgRNA靶位点。通过引物重叠PCR产生靶向嗜热分枝杆菌U6启动子驱动的sgRNA,并将其克隆到pJET1.2/blunt克隆载体中,该载体产生相应的质粒U6p-amdS sgRNA,U6p-cre-1-sgRNA和U6p-Mtclr-2-sgRNA。为了构建基因缺失底物,从p0380-neo质粒中扩增了PtrpC-neo盒。通过使用配对引物的PCR,从嗜热支原体基因组DNA中分别扩增出Mtclr-2的5’和3’个侧翼片段。使用NEB Gibson assem bly试剂盒将扩增的5’、3’和PtrpC-neo片段组装并连接到pJET1.2/blunt克隆载体中,以产生供体-clr-2 DNA序列。
参考文献:Zhang C, Li N, Rao L, Li J, Liu Q, Tian C. Development of an Efficient C-to-T Base-Editing System and Its Application to Cellulase Transcription Factor Precise Engineering in Thermophilic Fungus Myceliophthora thermophila. Microbiol Spectr. 2022 Jun 29;10(3):e0232121.
技术服务 Technology Services
联系我们
CONTACT US
-
0574-87917803
服务热线 -
testobio@163.com
邮箱
使用NEB Gibson组装试剂盒将UGI,Ptef1启动子(8)和TtrpC终止子扩增并组装到p0380-bar质粒(69)中,以形成evo-BE4max,GAM-BE4max和evo-CDA1编辑器。先前构建了Cas9表达PCR盒bar-Ptef1-Cas9-TtprC,以及选择标记neo和bar。为了选择靶向AMD,cre-1和Mtclr-2的特定sgRNA,使用sgRNACas9工具鉴定嗜热支原体基因组中的所有sgRNA靶位点,并选择高分的sgRNA靶位点。通过引物重叠PCR产生靶向嗜热分枝杆菌U6启动子驱动的sgRNA,并将其克隆到pJET1.2/blunt克隆载体中,该载体产生相应的质粒U6p-amdS sgRNA,U6p-cre-1-sgRNA和U6p-Mtclr-2-sgRNA。为了构建基因缺失底物,从p0380-neo质粒中扩增了PtrpC-neo盒。通过使用配对引物的PCR,从嗜热支原体基因组DNA中分别扩增出Mtclr-2的5’和3’个侧翼片段。使用NEB Gibson assem bly试剂盒将扩增的5’、3’和PtrpC-neo片段组装并连接到pJET1.2/blunt克隆载体中,以产生供体-clr-2 DNA序列。

参考文献:Zhang C, Li N, Rao L, Li J, Liu Q, Tian C. Development of an Efficient C-to-T Base-Editing System and Its Application to Cellulase Transcription Factor Precise Engineering in Thermophilic Fungus Myceliophthora thermophila. Microbiol Spectr. 2022 Jun 29;10(3):e0232121.







