设计了nagA和gamA基因的敲除盒,以阻断宿主菌株谷氨酸梭菌降解GlcNAc的途径。为了提高敲除效率,前部和后部区域的长度设计为约1000 bp。以谷氨酸杆菌S9114基因组序列为模板,PCR扩增nagA-gaA基因的同源臂片段na gaU和na gaD。使用凝胶回收试剂盒回收DNA片段。以携带Kana抗性基因片段lox-kan-loxp盒的质粒pDTW202为模板,扩增lox-kan-loxp盒。使用质粒提取试剂盒提取大肠杆菌衍生的pBluscriptII-SK(+)质粒,并测定DNA浓度。用XhoI和XbaI消化片段na gaU,用XbaI和BamH1消化片段lox-kan-loxp盒,用BamHI和EcoRI消化片段na gaD,用XhoⅠ和EcoRI酶切质粒pBluscriptII-SK(+)。使用凝胶回收试剂盒回收所有消化片段。使用T4 DNA连接酶以1:3的摩尔比(质粒:片段)将酶纯化的片段na-gaU、na-gaD和lox-kan-loxp盒连接到pBluscriptII SK(+)质粒上。然后将连接物转化到感受态大肠杆菌JM109细胞中,并接种到Kana抗性LB板上进行筛选。得到的正确转化体携带质粒pDTW NG,该质粒携带nagA/gaA敲除盒。将正确测序的pDTW-NG质粒转化到谷氨酸杆菌S9114菌株中。获得的重组基因组携带lox-kan-loxp盒片段,Kana抗性基因两端的lox序列可以被Cre酶特异性识别和切割。将携带Cre基因的质粒pDTW-109进一步电穿孔到突变菌株中。除了携带Cre基因外,该质粒还携带温度敏感复制子和氯霉素抗性基因。将含有卡那霉素和氯霉素的LBB板分别在25°C下划线,选择仅在氯霉素板上生长的转化体,并在37°C的培养箱中进一步培养,以去除pDTW-109质粒。使用与nagA和gamA基因相同的方法敲除ldh基因。
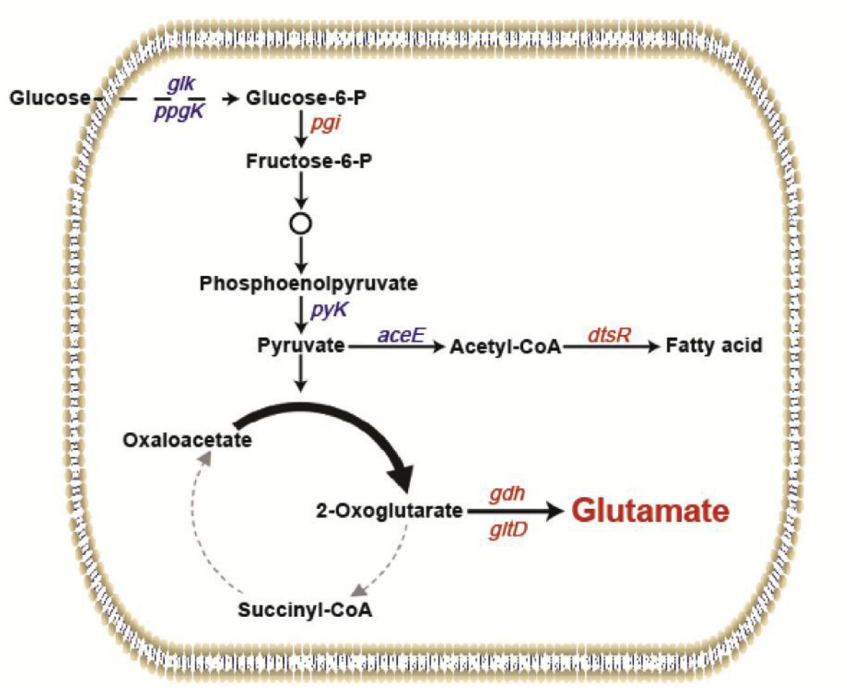
参考文献:Deng C, Lv X, Liu Y, Li J, Lu W, Du G, Liu L. Metabolic engineering of Corynebacterium glutamicum S9114 based on whole-genome sequencing for efficient N-acetylglucosamine synthesis. Synth Syst Biotechnol. 2019 Jun 6;4(3):120-129.
技术服务 Technology Services
联系我们
CONTACT US
-
0574-87917803
服务热线 -
testobio@163.com
邮箱
设计了nagA和gamA基因的敲除盒,以阻断宿主菌株谷氨酸梭菌降解GlcNAc的途径。为了提高敲除效率,前部和后部区域的长度设计为约1000 bp。以谷氨酸杆菌S9114基因组序列为模板,PCR扩增nagA-gaA基因的同源臂片段na gaU和na gaD。使用凝胶回收试剂盒回收DNA片段。以携带Kana抗性基因片段lox-kan-loxp盒的质粒pDTW202为模板,扩增lox-kan-loxp盒。使用质粒提取试剂盒提取大肠杆菌衍生的pBluscriptII-SK(+)质粒,并测定DNA浓度。用XhoI和XbaI消化片段na gaU,用XbaI和BamH1消化片段lox-kan-loxp盒,用BamHI和EcoRI消化片段na gaD,用XhoⅠ和EcoRI酶切质粒pBluscriptII-SK(+)。使用凝胶回收试剂盒回收所有消化片段。使用T4 DNA连接酶以1:3的摩尔比(质粒:片段)将酶纯化的片段na-gaU、na-gaD和lox-kan-loxp盒连接到pBluscriptII SK(+)质粒上。然后将连接物转化到感受态大肠杆菌JM109细胞中,并接种到Kana抗性LB板上进行筛选。得到的正确转化体携带质粒pDTW NG,该质粒携带nagA/gaA敲除盒。将正确测序的pDTW-NG质粒转化到谷氨酸杆菌S9114菌株中。获得的重组基因组携带lox-kan-loxp盒片段,Kana抗性基因两端的lox序列可以被Cre酶特异性识别和切割。将携带Cre基因的质粒pDTW-109进一步电穿孔到突变菌株中。除了携带Cre基因外,该质粒还携带温度敏感复制子和氯霉素抗性基因。将含有卡那霉素和氯霉素的LBB板分别在25°C下划线,选择仅在氯霉素板上生长的转化体,并在37°C的培养箱中进一步培养,以去除pDTW-109质粒。使用与nagA和gamA基因相同的方法敲除ldh基因。

参考文献:Deng C, Lv X, Liu Y, Li J, Lu W, Du G, Liu L. Metabolic engineering of Corynebacterium glutamicum S9114 based on whole-genome sequencing for efficient N-acetylglucosamine synthesis. Synth Syst Biotechnol. 2019 Jun 6;4(3):120-129.







