在罗伊氏乳杆菌中鉴定了一个II-A CRISPR-Cas9系统,并发现内源性Cas9(LrCas9)识别广泛的原间隔邻近基序(PAM)序列。我们对LrCas9进行了重编程,以获得高效的基因缺失(95.46%)、点突变(86.36%)、大片段缺失(40 kb)和基因整合(1743 bp,73.9%),揭示了黏液结合蛋白中重复保守结构域的功能。并通过基因缺失、点突变和基因整合,成功地验证和优化了内源性LrCas9的功能
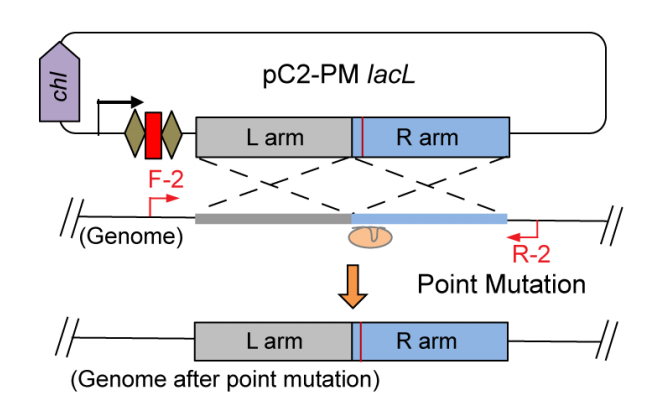
参考文献:Guo Q, Yan Y, Zhang Z, Xu B, Bangash HL, Sui X, Yang Y, Zhou Z, Zhao S, Peng N. Developing the Limosilactobacillus reuteri Chassis through an Endogenous Programmable Endonuclease-Based Genome Editing Tool. ACS Synth Biol. 2023 Nov 17;12(11):3487-3496.
技术服务 Technology Services
联系我们
CONTACT US
-
0574-87917803
服务热线 -
testobio@163.com
邮箱
在罗伊氏乳杆菌中鉴定了一个II-A CRISPR-Cas9系统,并发现内源性Cas9(LrCas9)识别广泛的原间隔邻近基序(PAM)序列。我们对LrCas9进行了重编程,以获得高效的基因缺失(95.46%)、点突变(86.36%)、大片段缺失(40 kb)和基因整合(1743 bp,73.9%),揭示了黏液结合蛋白中重复保守结构域的功能。并通过基因缺失、点突变和基因整合,成功地验证和优化了内源性LrCas9的功能

参考文献:Guo Q, Yan Y, Zhang Z, Xu B, Bangash HL, Sui X, Yang Y, Zhou Z, Zhao S, Peng N. Developing the Limosilactobacillus reuteri Chassis through an Endogenous Programmable Endonuclease-Based Genome Editing Tool. ACS Synth Biol. 2023 Nov 17;12(11):3487-3496.







