用ClaI对pUL5000进行切割获得含有gehC的片段并克隆到pBluescriptKS(+)中,形成pUL5010。用TaqI从pE194中切除出红霉素耐药基因盒(ermC)然后插入pUL5010的HinlI位点,形成pUL5011。为了使质粒在葡萄球菌中复制,将pCW59的HindIII标记的片段连接到pUL5011的EcoRI位点,构建了穿梭载体pUL5012。为了筛选成功转化的菌株,将含有被破坏的脂肪酶基因的穿梭载体pUL5012和一个不相容的质粒pTS01共转化到表皮葡萄球菌中。
gehD的失活原理与gehC相同,通过将含有ermC盒的pE194的SalI-XhoI片段插入到pUL5041的SalI位点,形成pUL5046。然后将合成的载体pUL5046连接到pCW59的HindIII片段上,得到穿梭载体pUL5047。
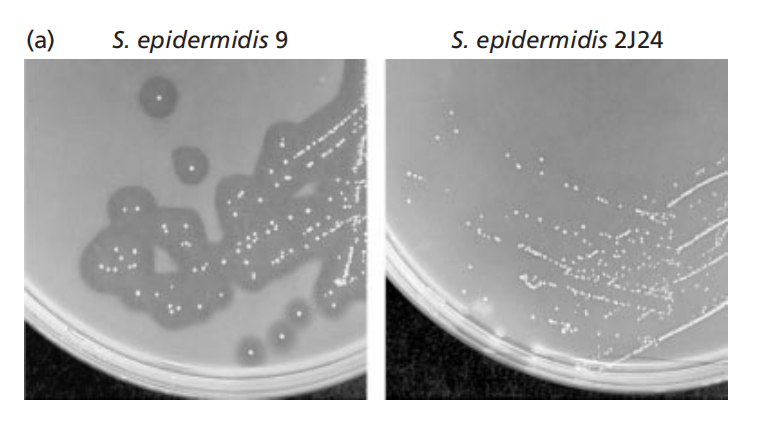
参考文献:Longshaw CM, Farrell AM, Wright JD, Holland KT. Identification of a second lipase gene, gehD, in Staphylococcus epidermidis: comparison of sequence with those of other staphylococcal lipases. Microbiology (Reading). 2000 Jun;146 ( Pt 6):1419-1427.
技术服务 Technology Services
联系我们
CONTACT US
-
0574-87917803
服务热线 -
testobio@163.com
邮箱
用ClaI对pUL5000进行切割获得含有gehC的片段并克隆到pBluescriptKS(+)中,形成pUL5010。用TaqI从pE194中切除出红霉素耐药基因盒(ermC)然后插入pUL5010的HinlI位点,形成pUL5011。为了使质粒在葡萄球菌中复制,将pCW59的HindIII标记的片段连接到pUL5011的EcoRI位点,构建了穿梭载体pUL5012。为了筛选成功转化的菌株,将含有被破坏的脂肪酶基因的穿梭载体pUL5012和一个不相容的质粒pTS01共转化到表皮葡萄球菌中。
gehD的失活原理与gehC相同,通过将含有ermC盒的pE194的SalI-XhoI片段插入到pUL5041的SalI位点,形成pUL5046。然后将合成的载体pUL5046连接到pCW59的HindIII片段上,得到穿梭载体pUL5047。

参考文献:Longshaw CM, Farrell AM, Wright JD, Holland KT. Identification of a second lipase gene, gehD, in Staphylococcus epidermidis: comparison of sequence with those of other staphylococcal lipases. Microbiology (Reading). 2000 Jun;146 ( Pt 6):1419-1427.







